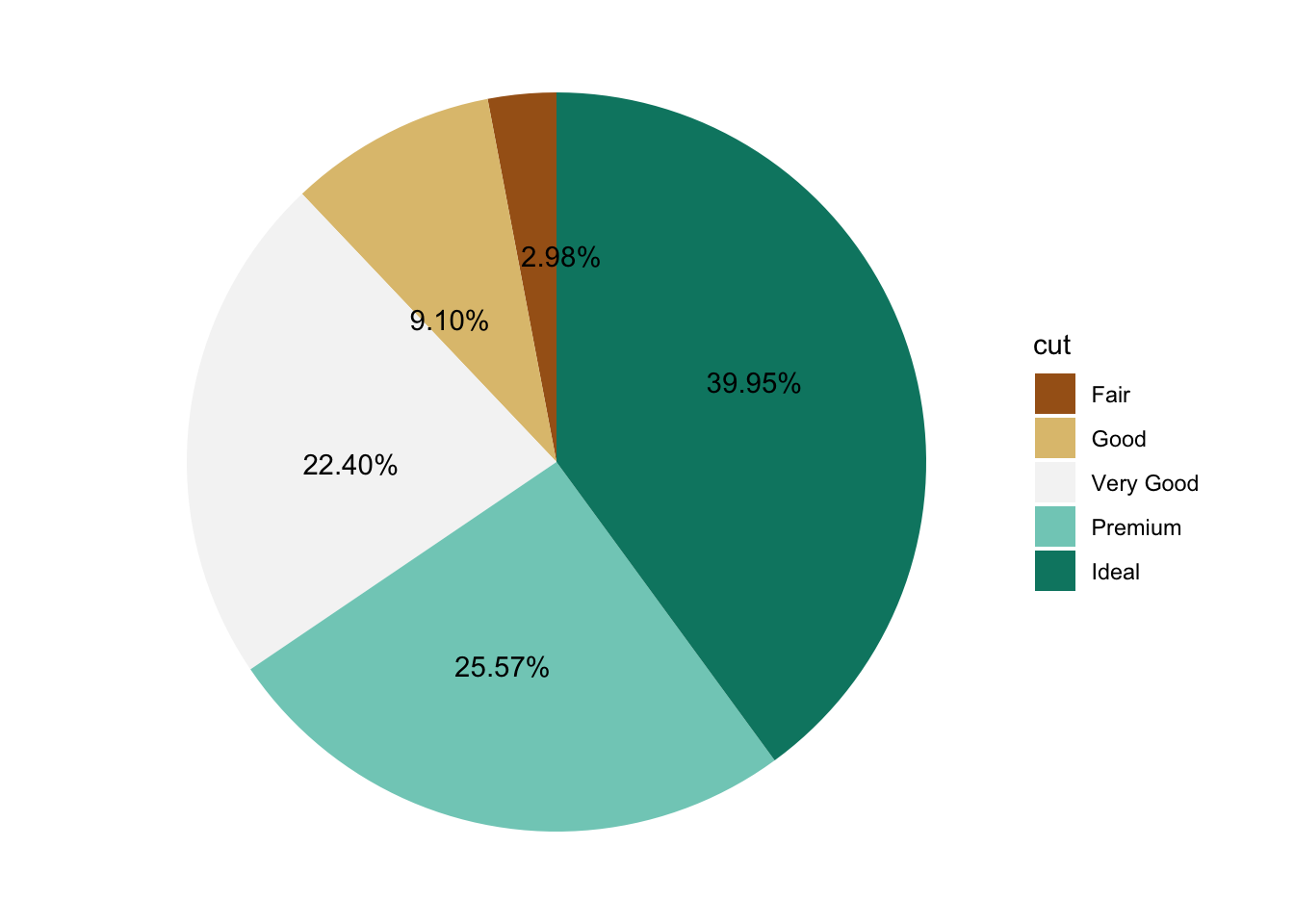
ggplot2 画百分比饼图
首先通过 count() 计算出各种 cut 的钻石各有多少个,存到名为 n 的列里,然后通过 mutate 计算出总和为1的占比。此时如果用 geom_col() 画图可以得出一个高度为1的堆积图。
library(tidyverse)
diamonds %>%
count(cut) %>%
mutate(p = n / sum(n)) %>%
ggplot(aes("", p, fill = cut)) +
geom_col() +
scale_fill_brewer(type = "div")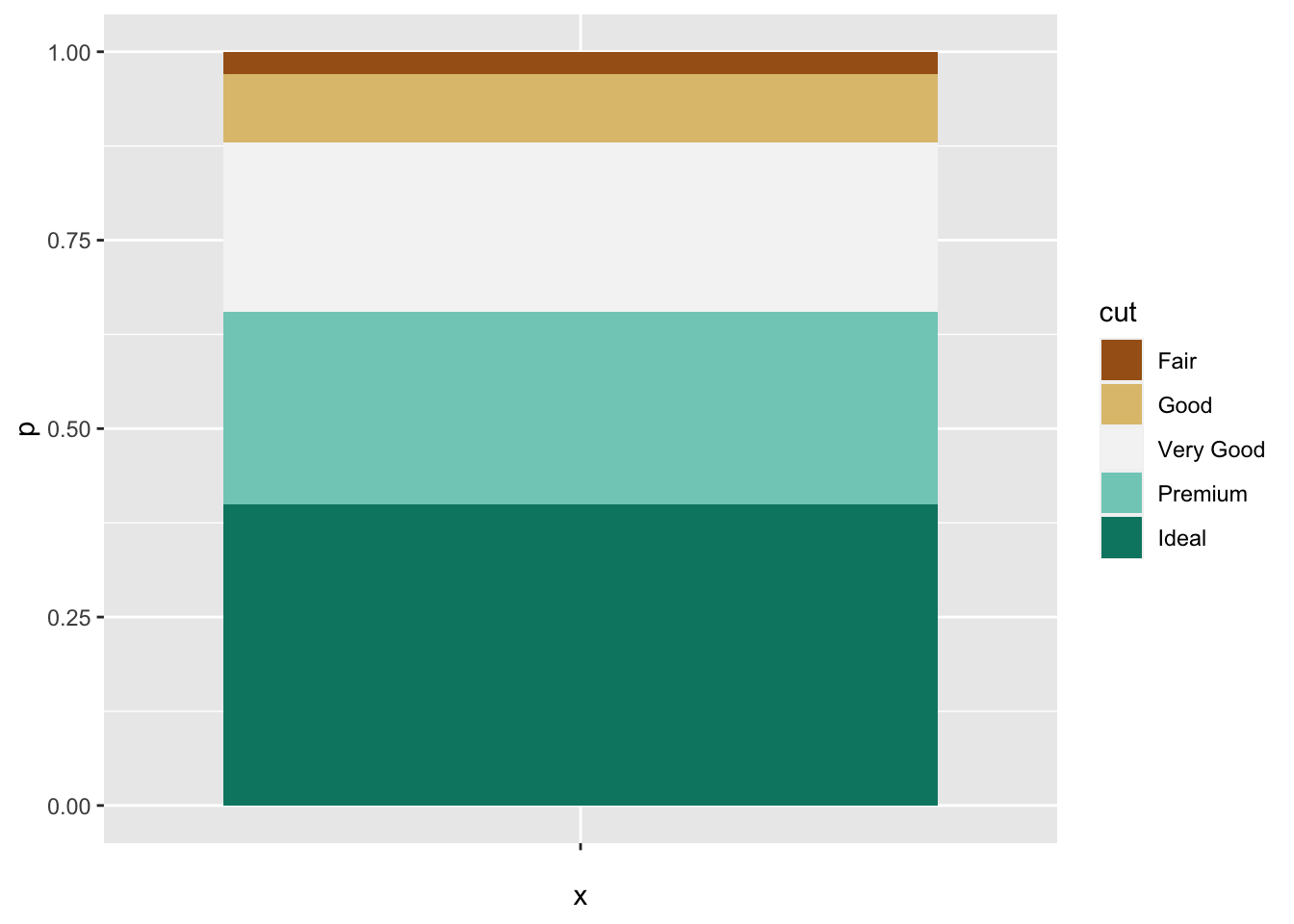
再加以 coord_polar() 变换转换为极座标,就可以画出一张饼图了。在此基础上多叠加一层 geom_text() 就实现了百分比的显示,非常灵活。
diamonds %>%
count(cut) %>%
mutate(p = n / sum(n)) %>%
mutate(labels = scales::label_percent(accuracy = 0.01)(n / sum(n))) %>%
ggplot(aes("", p, fill = cut)) +
geom_col() +
geom_text(aes(label = labels), position = position_stack(0.5)) +
coord_polar("y") +
scale_fill_brewer(type = "div")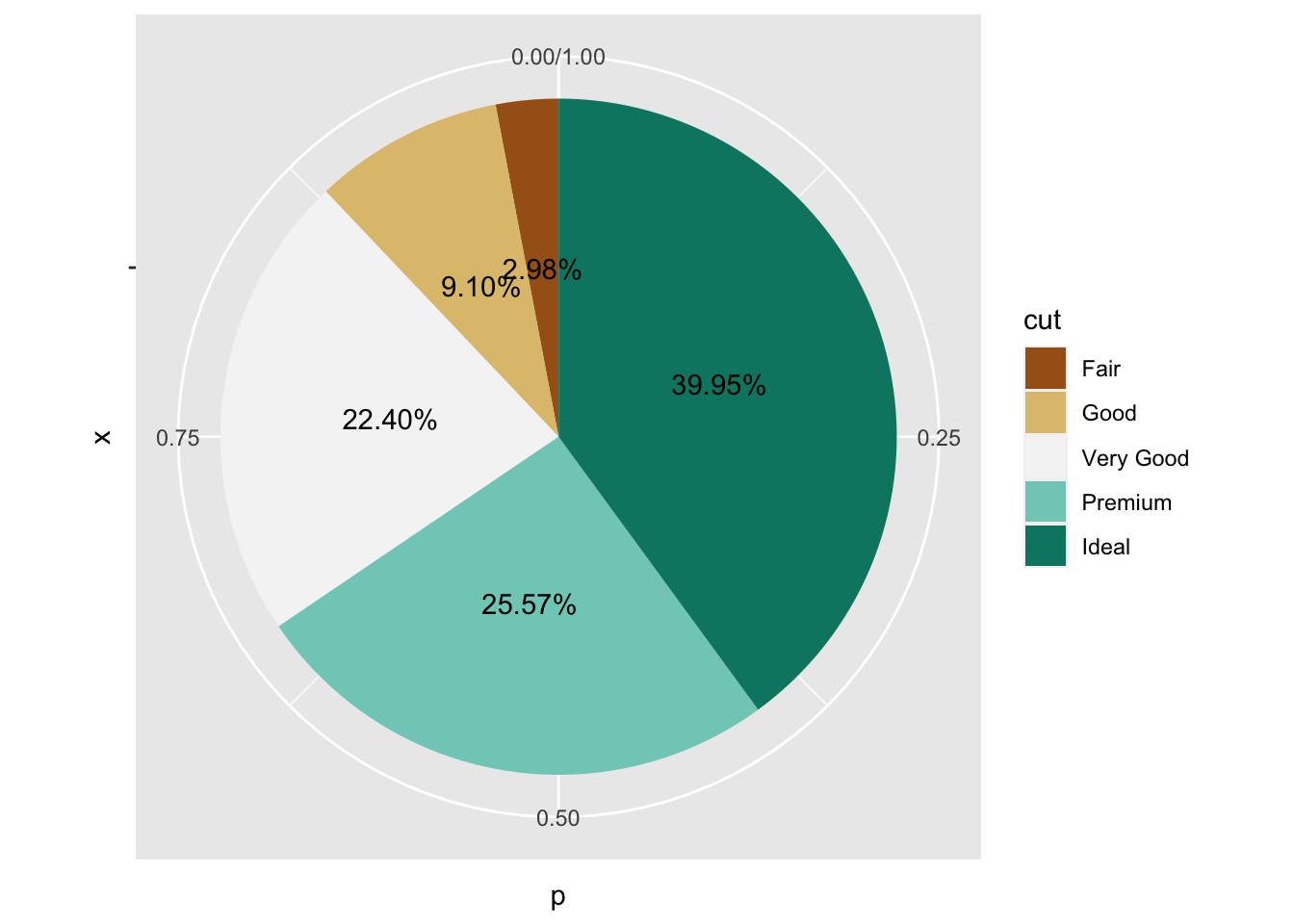
如果不想显示极座标轴和灰色背景,可以通过加上 theme_void() 来实现。对于重叠的文字则是可以用 ggrepel 打散,最终效果如图。